Spécifications générales¶
Révision du document de anne¶
| Date | |
| algorithme_15_08.doc | calcul moyenne et ecart type des beta i |
| 29/07/2010 | les genotypes manquants et option tirage ou non de PI |
| 07/06/2010 | creation |
Spécifications détaillées¶
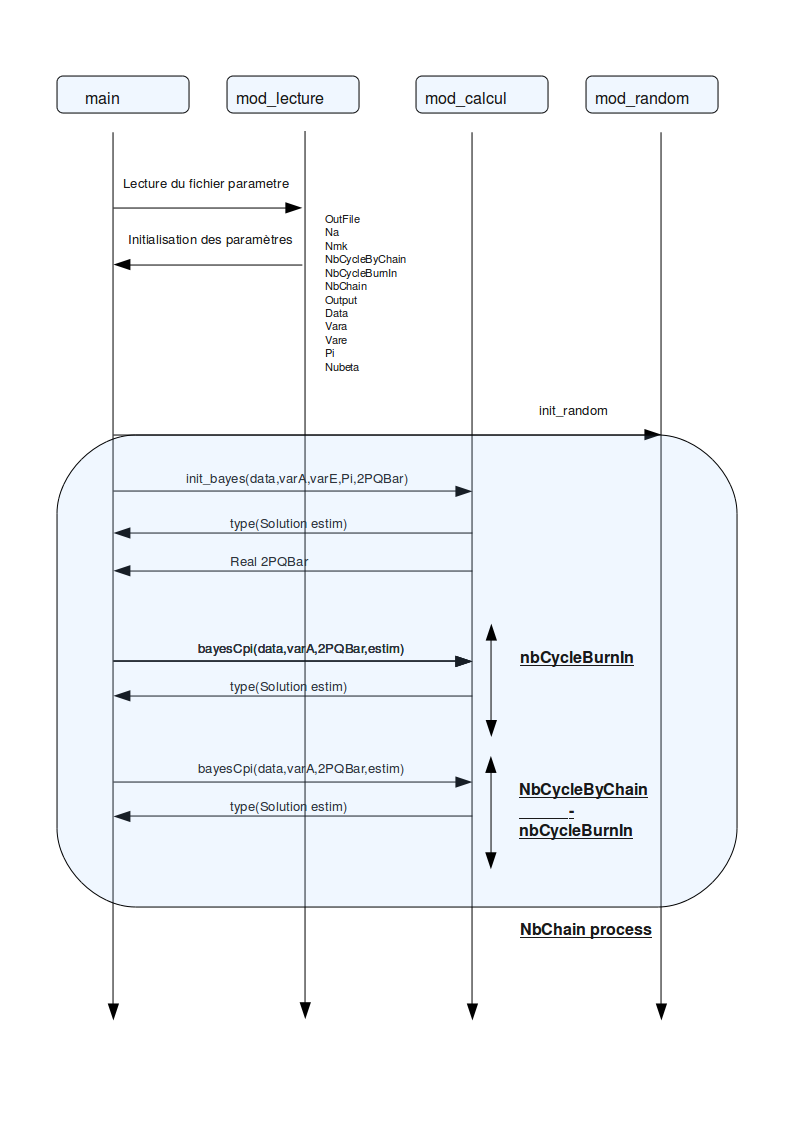
Description des modules¶
Les sources se trouvent dans le répertoire src. Il y a 6 modules (5 fortran, 1 C++) et un programme principale :
- main.f95 : programme principale exécutant plusieurs processus (nombre de chaine)
- mod_struct.f95 : module contenant les constantes et les structures de données du programme
- mod_lecture.f95 : routines pour la lecture des données
- mod_random.f95 : interface d'appel des routines de génération aléatoire dans des lois CHI2,Normal,Uniforme et beta
- mod_calcul.f95 : algorithme du bayes C pi
Structure de données¶
Type DATASET
Description du jeu de donnees¶
- na : nombre d'animaux
- nmk : nombre de marqueurs
- y : vecteur des performances (dimension : na)
- x : vecteur de typage 0 pour aa 1 pour Aa 2 pour AA (dimension : na,nmk)
- availXAnim : tableau de booleen (dimension na,nmk) pour la presence d un genotype d'un individu au marqueur
- availXProb : tableau de booleen (dimension nmk) pour la presence d'au moin un genotype manquant au marqueur
- probGenotypeCase : probabilite d'obtenir un genotype -1, 0 ou 1.
probGenotypeCase est calculé seulement si availXProb est a faux au marqueur i.
Type Solution
Description de l'estimation / solution¶
- mu : moyenne generale
- beta : vecteur des effets (dimension : nmk)
- deltaOne : (dimension : nmk) : vecteur de presence de l effet (1 ou 0)
- vare : variance residuelle
- varbeta : variance des effets SNP
- pi : pi
- nbloci : nombre de locus pris en compte (somme des delat i)
Type DistributionParameter
Stockage des solutions.
- Nb : nombre de points
- allVare : liste des variances residuelles.
- AllVarbeta : liste des variances genetiques
- allPi : liste des valeurs de Pi