Flow d'exécution du programme¶
Cette section décrit l'appel des fonctions sollicitées lors d'une analyse.
Déroulement du programme principale¶
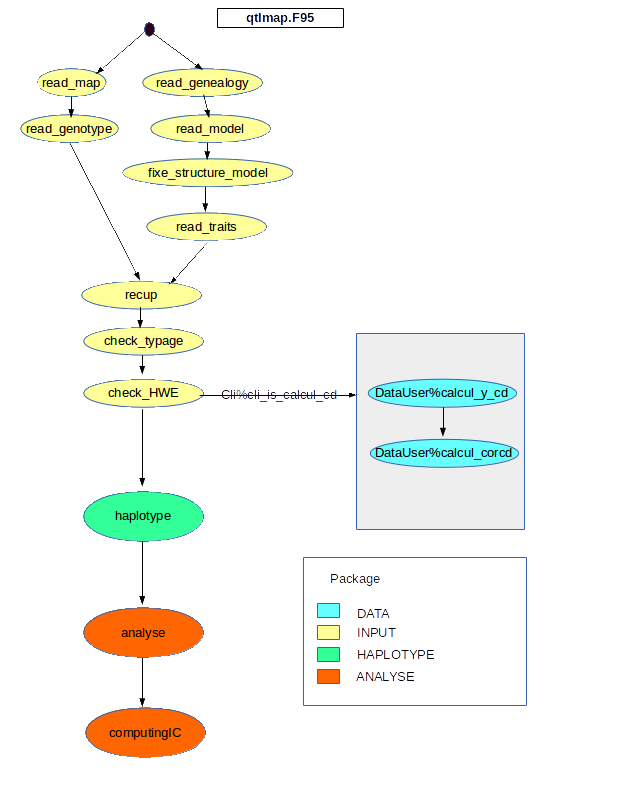
L'exécution d'une analyse sans simulation est décrite dans le diagramme suivant.
La première partie correspond à la lecture des données ainsi qu'a la vérification des génotypes. C'est le package INPUT qui est responsable de ces traitements.
Il existe un traitement spécifique si l'utilisateur demande de calculer les cd et les corrélations des cd (option --calcul-cd). Ce mécanisme a été mis en place lors de l’implémentation
du multicaractère de la thèse de Mohamed Kileh wais. Ce mecanisme peut etre appelé pour les autres analyses.
Le programme principal utilise succésivement les packages INPUT, HAPLOTYPE puis ANALYSE
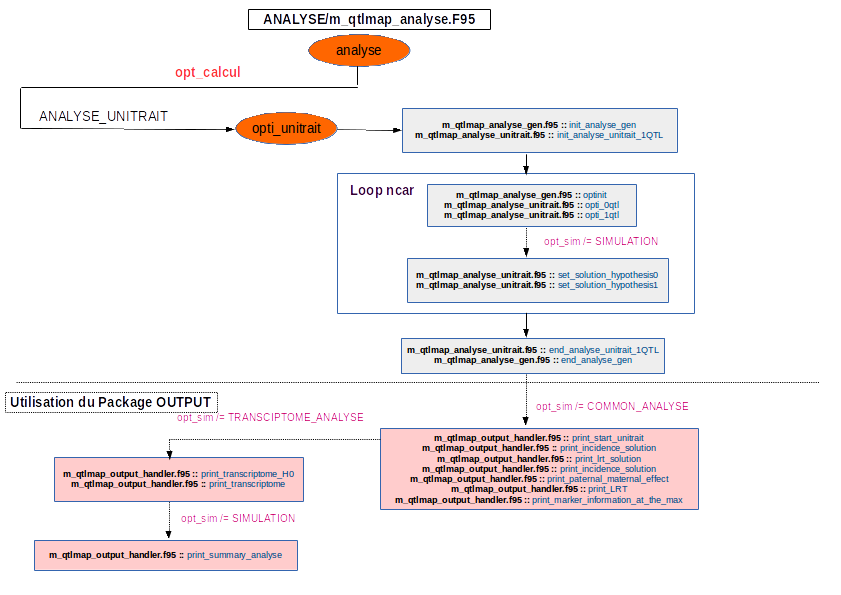
Le package ANALYSE¶
Les analyses modlin et dérivées¶
Voici le fonctionnement du calcul=1. Ce fonctionnement est le même pour les analyses suivantes :
| ID analyse | m_qtlmap_analyse.F95:: | utilisation du module |
| ANALYSE_UNITRAIT_MODLIN | opti_unitrait_modlin | m_qtlmap_analyse_modlin.f95 |
| ANALYSE_UNITRAIT_LDLA | opti_unitrait_modlin_new | m_qtlmap_analyse_modlin_ldla.f95 |
| ANALYSE_UNITRAIT_LD | opti_unitrait_modlin_new | m_qtlmap_analyse_modlin_ldla.f95 |
| ANALYSE_UNITRAIT_LA | opti_unitrait_modlin_new | m_qtlmap_analyse_modlin_ldla.f95 |
| ANALYSE_UNITRAIT_LDJH | opti_unitrait_modlin_new | m_qtlmap_analyse_modlin_ldla.f95 |
| ANALYSE_MULTITRAIT_DA | opti_multitrait_DA | m_qtlmap_analyse_multitrait_DA.f95 |
| ANALYSE_UNITRAIT_MODLIN_cox | opti_unitrait_modlin_cox | m_qtlmap_analyse_modlin_cox.f95 |
| ANALYSE_MULTITRAIT | opti_multitraits | m_qtlmap_analyse_multitrait.f95 |
Les analyses du module m_qtlmap_contingence.F95¶
Les analyses implémentées dans le module m_qtlmap_incidence.F95 ont un flow d'exécution différent des analyses de type modlin.
Ces analyses utilisent les même mécanismes pour la résolution d'un modèle. On les différencie par :
- le type de variance du modèle (homoscedastique et heteroscedastique)
- le modèle polygénique ou utilisant une matrice de parenté (modèle animal)
- la résolution du modèle (linéaire ou par l'optimisation de la fonction de vraisemblance)
- La construction de la matrice de contingence (les effets à estimer : moyenne générale, effets polygéniques, qtl, haplotypiques, de nuisance, caracteres en tant que covariable (these xiao), interaction,...)
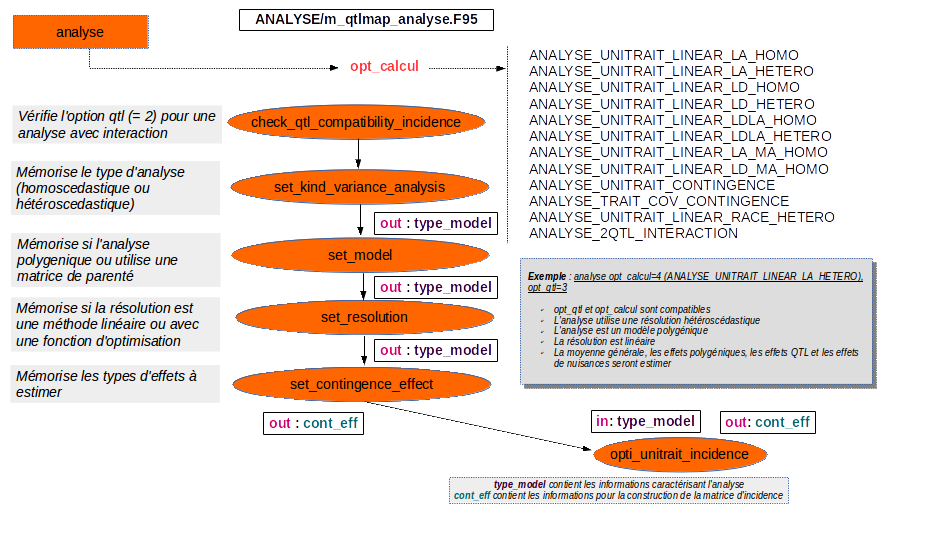
la variable type_model est un entier synthétisant une partie de l'information. Des constantes sont définit dans le module m_qtlmap_incidence.F95.
integer , parameter ,public :: MASK_INIT_MOD_INC = INT(b'0000000000000000') !//Exclusifs integer , parameter ,public :: KIND_HOMOSCEDASTIC = INT(b'0001000000000000') integer , parameter ,public :: KIND_HETEROSCEDASTIC = INT(b'0010000000000000') !//Exclusifs integer , parameter ,public :: MODEL_UNITRAIT_POLYGENIC = INT(b'0000000100000000') integer , parameter ,public :: MODEL_MULTITRAIT_POLYGENIC = INT(b'0000001000000000') integer , parameter ,public :: MODEL_MULTITRAIT_POLYGENIC_LU = INT(b'0000010000000000') integer , parameter ,public :: MODEL_UNITRAIT_RELATIONSHIP = INT(b'0000100000000000') !//Exclusifs integer , parameter ,public :: RESOLUTION_LINEAR = INT(b'0000000000010000') integer , parameter ,public :: RESOLUTION_OPTIMISATION = INT(b'0000000000100000')
une subroutine set_parameter est utilisée pour mettre à jour la variable type_model (m_qtlmap_incidence.F95)
subroutine set_parameter(newparameter,allparameter) integer , intent(in) :: newparameter integer , intent(inout) :: allparameter allparameter = ior(newparameter,allparameter) !// ou logic bit à bit pour les caracteristiques de l'analyse courante (homoscedastic,hetero,lineaire,optim,model animal,....) end subroutine set_parameter function is_parameter(testparam,allparameter) result(res) integer , intent(in) :: testparam,allparameter logical :: res res = iand(testparam,allparameter) /= MASK_INIT_MOD_INC end function
La variable cont_eff est de type type_effect_contingence (m_qtlmap_incidence.F95). On assigne l'ordre des effets à estimer (de 1 à N) et d'insertion dans la matrice de contingence.
La valeur 0 signifie que l'effet n'est pas à estimer.
type type_effect_contingence integer :: general = 1 integer :: polygenic = 2 integer :: qtl = 3 integer :: haplo = 0 integer :: nuis = 4 integer :: traits = 0 integer :: inter = 0 integer :: nbeff = 7 ! have to be inscrease if other kind of effects end type type_effect_contingence
Plusieurs constantes de ce type sont définit dans le module m_qtlmap_incidence.F95 pour les différentes analyses qui sont implémentées le module.
type(type_effect_contingence) ,parameter :: CONTINGENCE_QTL = type_effect_contingence(1,3,2,0,4,0,0) type(type_effect_contingence) ,parameter :: CONTINGENCE_HAPLO = type_effect_contingence(1,3,0,2,4,0,0) type(type_effect_contingence) ,parameter :: CONTINGENCE_HAPLO_QTL = type_effect_contingence(1,4,3,2,5,0,0) type(type_effect_contingence) ,parameter :: CONTINGENCE_QTL_TRAITS = type_effect_contingence(1,3,2,0,4,5,0) type(type_effect_contingence) ,parameter :: CONTINGENCE_QTL_INTER = type_effect_contingence(1,5,2,0,4,0,3) type(type_effect_contingence) ,parameter :: CONTINGENCE_QTL_WITHOUT_POLY = type_effect_contingence(1,0,2,0,3,0,0) type(type_effect_contingence) ,parameter :: CONTINGENCE_HAPLO_WITHOUT_POLY = type_effect_contingence(1,0,0,2,3,0,0) type(type_effect_contingence) ,parameter :: CONTINGENCE_RACE = type_effect_contingence(1,3,2,0,4,0,0)