Modele Animal¶
Développement du modèle¶
Le développement de cet analyse à été réalisé à partir du module m_qtlmap_incidence.F95 et se base sur les développements qui ont été réalisé pour la linéarisation des analyses MODLIN.
2 sources sont impactés :
- m_qtlmap_incidence.F95
- m_qtlmap_incidence_linear.f95
- cuda_qtlmap_linear.cu (implémentation GPU de la méthode)
Modèle¶
sachant:
M = (I - (I + lambda*A^-1)
On calcul la solution b :
bHat = (X'.M.X)^-1 . X'.M.Y
Description des subroutines¶
m_qtlmap_incidence.f95/build_M_modanim¶
Cette routine calcul l'entité
M = (I - (I + lambda*A^-1)
m_qtlmap_incidence.f95/build_A_modanim¶
Construction de la matrice de parenté en utilisant la méthode récursive de Henderson
A = L.L' Aij = SUM k=1 ( Lik.Ljk ) i>j : Lij = (Aij - Sum Lik.Ljk) / Ljj i==j : Lii = SQRT(Aii - SUM Lik^2)
elle est appelé par la subroutine build_M_modanim
m_qtlmap_incidence_linear.f95/modele_MA_homoscedastic¶
Calcul et estimation des paramètres sur chaque position testée.
- Calcul de
X'.M. X-1 - Décomposition de cholesky
- Calcul de
X'.M.Y - Calcul de la variance résiduelle
Test du Modèle Animal¶
Attention ces scripts sont un developement spécifique et non robuste à une utilisation autre que le test du modèle animal
L'archive Workflow-ModAnimal.tar.gz contient un ensemble de script permettant de tester la puissance et la précision du modèle Animal dans QTLMap.
Ce test génère des simulations LDSO (avec un QTL unique) à partir d'un fichier pédigrée (ID PERE MERE GENERATION) puis éxécute les analyses QTLMap (Homoscedastic classic, Heteroscedastic classic, Modèle Animal).
Contenu de l'archive¶
- Un générateur de généalogie src/creat_pedig.f (compil.sh compile le programme et le met dans le répertoire bin)
- ./bin/ldso (l'executable ldso)
- ./utils/ un ensemble de script pour le workflow d'excécution
- functions.sh définition des fonctions utiliser dans le workflow
- simul_all.sh simule le jeux de données en appelant dans un premier temps create_pedig puis ldso
- workflow_ldso_qtlmap.sh : boucle sur
- simulation du jeux de donnees
- Execution des 3 analyses QTLMap
- execute_all.sh le script principale d’exécution du workflow
Configuration de la simulation¶
Le script execute_all.sh contient la configuration générale de la simulation :- Emplacement des executables LDSO,CREATEPEDIG,QTLMAP
- Paramètres de generation du fichier de généalogie :
- Paramètres de generation de la carte
- Options des analyses QTLMap
#!/bin/bash ROOT=$PWD CREATEPEDIG=$ROOT/bin/create_pedig LDSO=$ROOT/bin/ldso QTLMAP=/home/ofilangi/workspace-8/QTLMap-Types/build/cuda/src/qtlmap #TO generate only dataset and qtlmap file without execution.... ACTIVE_QTLMAP="OK" # "OK" ou "KO" # Nombre de simulation LDSO + QTLMAP NSIM=10 #Nombre de simulation QTLMAP NSIM_SEUIL_QTLMAP=100 LONG_CHROMO=1.0 NB_MARKER=200 POS_QTL=0.450 EFF_QTL=0.5 H2=0.3 RATIO_POLQTL=0.500 NG=5 # Nombre de generation NMPED=5 # Nombre de male NFMPED=3 # Nombre de femelle par pere NDFPED=20 # Nombre de descendant par femelle par generation historique NMQTL=5 # Nombre de male dans le protocole QTL NFMQTL=3 # Nombre de femelle par pere dans le protocole QTL NDFQTL=20 # Nombre de descendant par femelle par generation historique dans le protocole QTL QTLMAP_STEP=0.05 #analyse demi-frere QTLMAP_NDMIN=25 QTLMAP_H2=$H2 export OMP_NUM_THREADS=4 source $ROOT/utils/workflow_ldso_qtlmap.sh
la derniere ligne lance l'execution du workflow.
Résultats¶
Ce script génère des répertoire et fichiers préfixer par certain paramètre.
Dans le repertoire suivant :
NG5_NMPED5_NFMPED3_NDFPED20_NMQTL5_NFMQTL3_NDFQTL20/H2_0.3_RATIO_POLQTL0.500_LONGCHROMO1.0_NMK200_POS_QTL0.450_EFF_QTL0.5/
nous trouvons un certain nombre de répertoire contenant chacun une simulation (le nom des répertoire correspond au seed utiliser pour generer la simulation) ainsi que les fichiers
RESUME_CALCUL_NDMIN25_100_STEP0.05_MODELH2_0.3_NSIMTHRESH100_<NUM> suffixé par l'option --calcul de QTLMap
18290_18568_4346_9598 21245_7743_26746_15145 30845_3724_3597_10001 9068_7864_29322_27199 18501_18723_18088_27308 21511_18206_14195_6026 587_8406_23092_4305 18973_15872_4476_1008 25300_29409_9347_12659 5917_4652_2596_19926 RESUME_CALCUL_NDMIN25_100_STEP0.05_MODELH2_0.3_NSIMTHRESH100_29 RESUME_CALCUL_NDMIN25_100_STEP0.05_MODELH2_0.3_NSIMTHRESH100_3 RESUME_CALCUL_NDMIN25_100_STEP0.05_MODELH2_0.3_NSIMTHRESH100_4
Les fichier RESUME contiennent l'information sur le resultat de chaque simulation
== INFO DATASET SIMUL == LONG_CHROMO=1.0 NB_MARKER=200 POS_QTL=0.450 EFF_QTL=0.5 H2=0.3 RATIO_POLQTL=0.500 --------------- NG=5 NMPED=5 NFMPED=3 NDFPED=20 NMQTL=5 NFMQTL=3 NDFQTL=20 --------------- QTLMAP_H2=0.3 QTLMAP_NSIM=100 == SIMUL == GROUPE_SEED POSIQTL LRT MARK_FLK_LEFT MARK_FLK_RIGHT THRES_0.1000 THRES_0.0500 THRES_0.0100 THRES_0.0050 THRES_0.0027 THRES_0.0001 18290_18568_4346_9598 18.43 23.58 43.21 47.95 49.74 50.81 51.06 51.29 5917_4652_2596_19926 16.15 18.52 26.13 27.96 28.66 29.08 29.17 29.27 9068_7864_29322_27199 15.70 18.08 20.42 20.95 21.15 21.27 21.30 21.33 587_8406_23092_4305 0.5000 12.27086 SNP100 SNP101 19.14 22.58 40.71 46.06 48.13 49.38 49.67 49.95 18973_15872_4476_1008 0.4500 17.09385 SNP90 SNP92 21.09 25.28 53.33 60.95 63.89 65.65 66.07 66.45 18501_18723_18088_27308 0.4500 25.45973 SNP90 SNP92 20.00 23.09 28.13 28.60 28.75 28.84 28.86 28.87 21245_7743_26746_15145 0.3500 11.91909 SNP70 SNP71 16.69 21.45 32.61 33.45 33.71 33.84 33.87 33.90 21511_18206_14195_6026 0.4500 27.70589 SNP90 SNP92 16.54 19.04 23.21 23.66 23.82 23.91 23.93 23.95 25300_29409_9347_12659 0.4500 27.85610 SNP90 SNP92 16.75 21.14 29.66 30.58 30.90 31.07 31.11 31.15 30845_3724_3597_10001 0.6000 18.88219 SNP120 SNP121 19.82 22.22 24.77 25.02 25.10 25.15 25.16 25.17
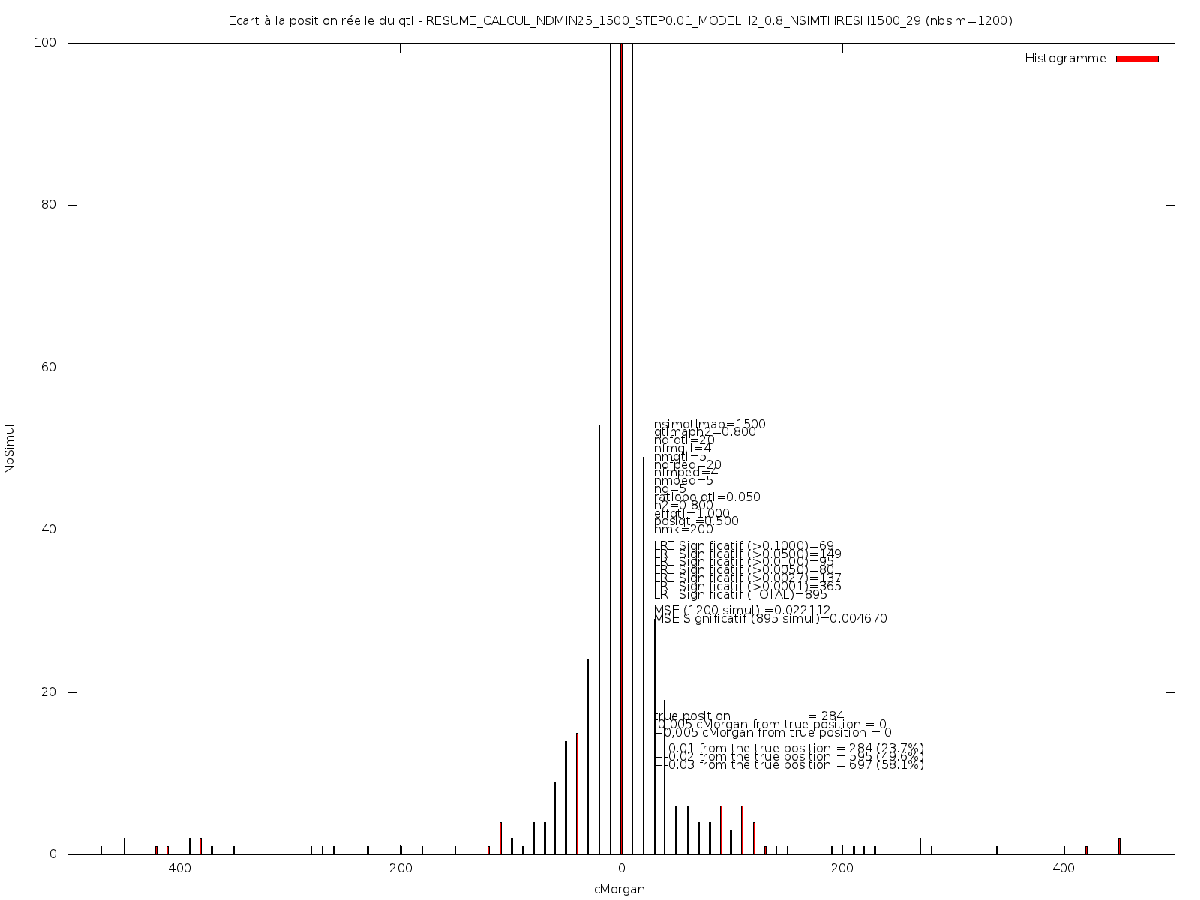
Information graphique¶
On peux obtenir une information graphique :
distribution des LRT Max de la méthode en executant le script ./utils/analyse_resume.sh <CHEMIN>/RESUME_CALCUL_NDMIN25_100_STEP0.05_MODELH2_0.3_NSIMTHRESH100_<NUM>
Ce script genere un repertoire RAPPORT contenant les graphes
Bench¶
| Date | Resultats | CR |
|---|---|---|
| 10 Avril 2012 | 120409-12_47_07.zip | CR 28-30 3 2012.docx |